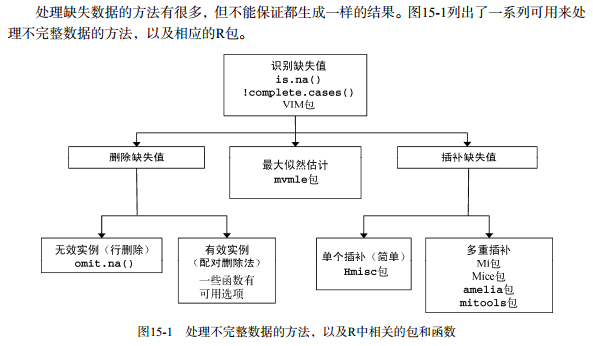
缺失值处理一般包括三步:
1. 识别缺失数据;
2. 检查导致数据缺失的原因;
3. 删除包含缺失值的实例或用合理的数值代替(插补)缺失值。
- 1.判断缺失值
函数is.na()、is.nan()和is.infinite()可分别用来识别缺失值、不可能值和无穷值。每个返回结果都是
TRUE或FALSE
na表示缺失值
nan表示NOT A NUMBER
infinite表示+-Inf
一定要亲手试x = 0/0,以及x = 1/0
>x <- NA
> is.na(x)
[1] TRUE
> is.nan(x)
[1] FALSE
> is.infinite(x)
[1] FALSE函数complete.cases()可用来识别矩阵或数据框中没有缺失值的行
超级好用
#加载数据集
>data(sleep,package = "VIM")
#没有缺失值的行
>sleep[complete.cases(sleep),]
#列出有一个或多个缺失值的行
>sleep[!complete.cases(sleep),] - 2.图形探究缺失数
#自己生成图形
>library("VIM")
>aggr(sleep,prop = FALSE,numbers = TRUE)matrixplot()函数可生成展示每个实例数据的图形
#自己生成图形
marginplot(sleep[c("Gest","Dream")],phc=c(20),col=c("darkgray","red","blue"))- 3.1删除缺失值
函数complete.cases()可以用来存储没有缺失值的数据框或者矩阵形式的实例(行):
#可以把mydata替换成sleep,延续上面的代码
>newdata <- mydata[complete.cases(mydata),]同样的结果可以用na.omit函数获得:
#可以把mydata替换成sleep,延续上面的代码
>newdata <- na.omit(mydata)两行代码表示的意思都是:mydata中所有包含缺失数据的行都被删除,然后结果才存储到
newdata中,以后拟合就用新数据newdata.
处理含缺失值的数据集时,成对删除常作为行删除的备选方法使用。对于成对删除,观测只
是当它含缺失数据的变量涉及某个特定分析时才会被删除:
>cor(sleep,use = "pairwise.complete.obs")3.2补全缺失值
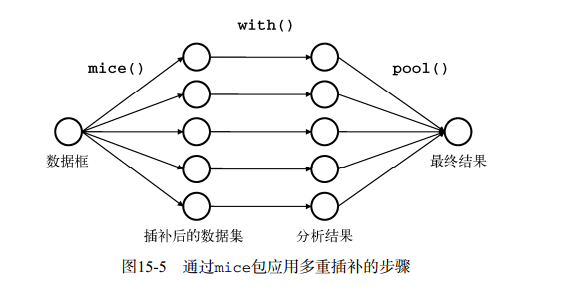
函数mice()首先从一个包含缺失数据的数据框开始,然后返回一个包含多个(默认为5个)完
整数据集的对象。每个完整数据集都是通过对原始数据框中的缺失数据进行插补而生成的。由于插
补有随机的成分,因此每个完整数据集都略有不同。然后,with()函数可依次对每个完整数据集
应用统计模型(如线性模型或广义线性模型),最 后 ,pool()函数将这些单独的分析结果整合为一
组结果。最终模型的标准误和p值都将准确地反映出由于缺失值和多重插补而产生的不确定性。
>library(mice)
>data("sleep",package = "VIM")
>imp <- mice(sleep,seed = 1234)
#imp <- mice(sleep)
>fit <- with(imp,lm(Dream~Span+Gest))
fit
>pooled <- pool(fit)
>summary(pooled)
imp
>dataset2 <- complete(imp,action = 2)
>cor(sleep,use = "pairwise.complete.obs")dataset2 is the newData
下图为完整缺失值处理图片:
来源:CSDN
作者:亓了个葩
链接:https://blog.csdn.net/skyonefly/article/details/52050005