注:本篇博客所有案例数据由MOOC平台提供!
无监督学习-聚类
K-means算法
k-means算法以k为参数,将n个对象分成k个簇,使簇内具有较高的相似度,而簇内具有较高的相似度,而簇间的相似度较低。
其处理过程如下:
(1)随机选择k个点作为初始的聚类中心;
(2)对于剩下的点,根据其与聚类中心的距离,将其归入最近的簇;
(3)对每个簇,计算所有点的均值作为新的聚类中心;
(4)重复(2)、(3)直到聚类中心不再发生变化。

import numpy as np
from sklearn.cluster import KMeans
def loadData(filePath):
fr = open(filePath,'r+')
lines = fr.readlines()
retData = []
retCityName = []
for line in lines:
items = line.strip().split(",")
retCityName.append(items[0])
retData.append([float(items[i]) for i in range(1,len(items))])
return retData,retCityName
if __name__ == '__main__':
data,cityName = loadData('city.txt')
km = KMeans(n_clusters=4)
label = km.fit_predict(data)
expenses = np.sum(km.cluster_centers_,axis=1)
#print(expenses)
CityCluster = [[],[],[],[]]
for i in range(len(cityName)):
CityCluster[label[i]].append(cityName[i])
for i in range(len(CityCluster)):
print("Expenses:%.2f" % expenses[i])
print(CityCluster[i])
输出结果:

DBSCAN密度聚类
DBSCAN算法是一种基于密度的聚类算法
特点:1.聚类的时候不需要预先指定簇的个数 2.最终的簇的个数不定
DBSCAN算法将数据点分为三类:
核心点:在半径Eps内含有超过MinPts数目的点
边界点:在半径Eps内的数量小于MinPts,但是落在核心点的邻域内
噪音点:既不是核心点也不是边界点的点
DBSCAN算法流程:
(1)将所有点标记为核心点、边界点和噪音点;
(2)删除噪音点;
(3)为距离在Eps之内的所有核心点之间赋予一条边;
(4)每组连通的核心点形成一个簇;
(5)将每个边界点指派为一个与之关联的核心点的簇中(哪一个核心点的半径范围之内)。

上网时间聚类:
import numpy as np
import sklearn.cluster as skc
from sklearn import metrics
import matplotlib.pyplot as plt
mac2id=dict()
onlinetimes=[]
f=open('TestData.txt',encoding='utf-8')
for line in f:
mac=line.split(',')[2]
onlinetime=int(line.split(',')[6])
starttime=int(line.split(',')[4].split(' ')[1].split(':')[0])
if mac not in mac2id:
mac2id[mac]=len(onlinetimes)
onlinetimes.append((starttime,onlinetime))
else:
onlinetimes[mac2id[mac]]=[(starttime,onlinetime)]
real_X=np.array(onlinetimes).reshape((-1,2))
X=real_X[:,0:1]
db=skc.DBSCAN(eps=0.01,min_samples=20).fit(X)
labels = db.labels_
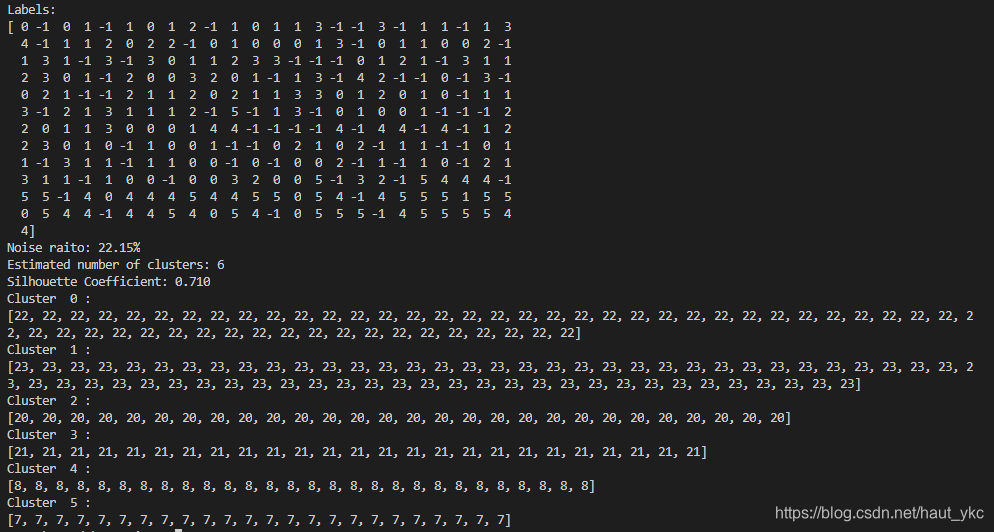
print('Labels:')
print(labels)
raito=len(labels[labels[:] == -1]) / len(labels)
print('Noise raito:',format(raito, '.2%'))
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0)
print('Estimated number of clusters: %d' % n_clusters_)
print("Silhouette Coefficient: %0.3f"% metrics.silhouette_score(X, labels))
for i in range(n_clusters_):
print('Cluster ',i,':')
print(list(X[labels == i].flatten()))
plt.hist(X,24)
输出结果:
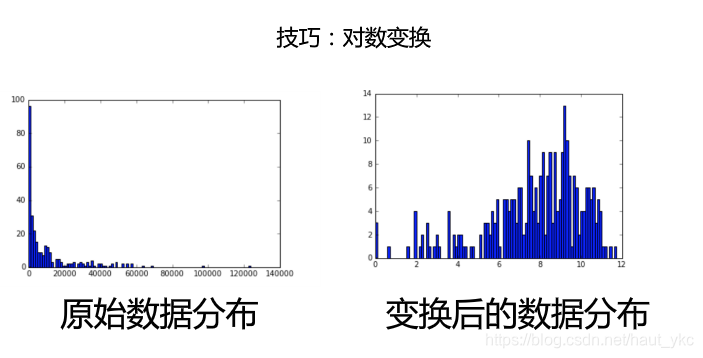
注意:若将数据可视化后原始数据分布不够明显,则可以对其进行对数变换。
无监督学习-降维
主成分分析(PCA)
主成分分析(Principal Component Analysis,PCA)是最常用的一种降维方法,通常用于高维数据集的探索与可视化,还可以用作数据压缩和预处理等。
PCA可以把具有相关性的高维变量合成为线性无关的低维变量,称为主成分。主成分能够尽可能保留原始数据的信息。
在介绍PCA原理之前需要回顾涉及到的相关术语:
方差:是各个样本和样本均值的差的平方和的均值,用来度量一组数据的分散程度。
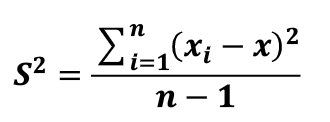
协方差:用于度量两个变量之间的线性相关程度,若两个变量的协方差为0,则可认为二者线性无关。协方差矩阵则是由变量的协方差值构成的矩阵(对称阵)。
![]()
特征向量:矩阵的特征向量是描述数据集结构的非零向量,并满足如下公式:
![]()

import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
from sklearn.datasets import load_iris
data = load_iris()
y = data.target
X = data.data
pca = PCA(n_components=2)
reduced_X = pca.fit_transform(X)
red_x, red_y = [], []
blue_x, blue_y = [], []
green_x, green_y = [], []
for i in range(len(reduced_X)):
if y[i] == 0:
red_x.append(reduced_X[i][0])
red_y.append(reduced_X[i][1])
elif y[i] == 1:
blue_x.append(reduced_X[i][0])
blue_y.append(reduced_X[i][1])
else:
green_x.append(reduced_X[i][0])
green_y.append(reduced_X[i][1])
plt.scatter(red_x, red_y, c='r', marker='x')
plt.scatter(blue_x, blue_y, c='b', marker='D')
plt.scatter(green_x, green_y, c='g', marker='.')
plt.show()
输出:
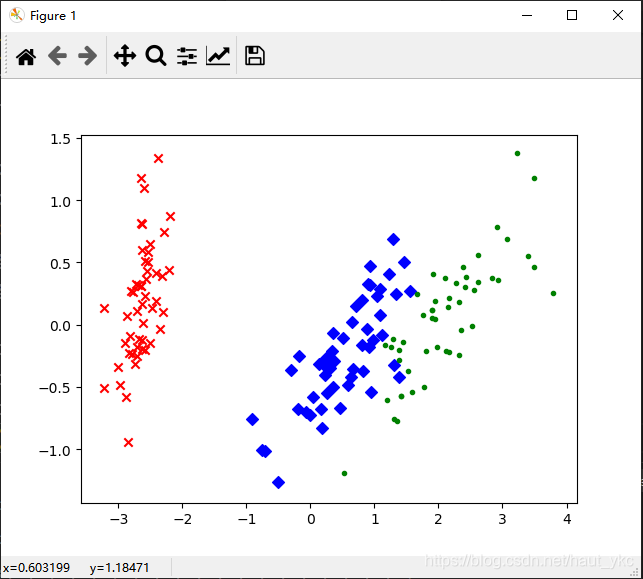
结果展示:可以看出,降维后数据仍能够清晰地分成三类。这样不仅能削减数据的维度,降低分类任务的工作量,还能保证分类的质量。
非负矩阵分解(NMF)
非负矩阵分解(Non-negative Matrix Factorization,NMF)是在矩阵中所有元素均为非负数约束条件之下的矩阵分解方法。
基本思想:给定一个非负矩阵V,NMF能够找到一个非负矩阵W和一个非负矩阵H,使得矩阵W和H的乘积近似等于V中的值。
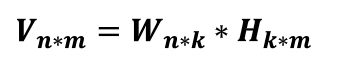
其中W矩阵是基础图像矩阵,相当于从原矩阵V中抽取出来的特征。H矩阵为系数矩阵。
因此NMF能够广泛应用于图像分析、文本挖掘和语言处理等领域。
矩阵分解优化目标:最小化W矩阵H矩阵的乘积和原始矩阵之间的差别。
![]()
基于KL散度的优化目标,损失函数如下:
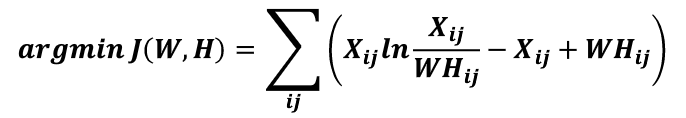
在sklearn库中,可以使用sklearn.decomposition.NMF加载NMF算法,主要参数有:
n_components:用于指定分解后矩阵的单个维度k;
init:W矩阵和H矩阵的初始化方式,默认为‘nndsvdar’。其他参数可以看官网API解释。


from numpy.random import RandomState
import matplotlib.pyplot as plt
from sklearn.datasets import fetch_olivetti_faces
from sklearn import decomposition
n_row, n_col = 2, 3
n_components = n_row * n_col
image_shape = (64, 64)
###############################################################################
# Load faces data
dataset = fetch_olivetti_faces(shuffle=True, random_state=RandomState(0))
faces = dataset.data
###############################################################################
def plot_gallery(title, images, n_col=n_col, n_row=n_row):
plt.figure(figsize=(2. * n_col, 2.26 * n_row))
plt.suptitle(title, size=16)
for i, comp in enumerate(images):
plt.subplot(n_row, n_col, i + 1)
vmax = max(comp.max(), -comp.min())
plt.imshow(comp.reshape(image_shape), cmap=plt.cm.gray,
interpolation='nearest', vmin=-vmax, vmax=vmax)
plt.xticks(())
plt.yticks(())
plt.subplots_adjust(0.01, 0.05, 0.99, 0.94, 0.04, 0.)
plot_gallery("First centered Olivetti faces", faces[:n_components])
###############################################################################
estimators = [
('Eigenfaces - PCA using randomized SVD',
decomposition.PCA(n_components=6,whiten=True)),
('Non-negative components - NMF',
decomposition.NMF(n_components=6, init='nndsvda', tol=5e-3))
]
###############################################################################
for name, estimator in estimators:
print("Extracting the top %d %s..." % (n_components, name))
print(faces.shape)
estimator.fit(faces)
components_ = estimator.components_
plot_gallery(name, components_[:n_components])
plt.show()
输出结果:



来源:CSDN
作者:袁珂晨
链接:https://blog.csdn.net/haut_ykc/article/details/103851502